W sierpniu 2019 roku zamówiliśmy w Instytucie Genetyki Sądowej w Bydgoszczy badania genealogiczne "w zakresie loci STR chromosomu Y" dla jednego z Piotrowskich ze Strachociny (z "klanu" Błaszczychów, mniejsza z tym kto to konkretnie).
Intencją zlecenia było uwiarygodnienie bądź też zanegowanie przekonania strachockich Piotrowskich o swoim pochodzeniu od Najman Bega, jednego z prominentnych tatarskich uczestników Bitwy pod Grunwaldem (O zasadności takiego przekonania piszemy w dostępnym w naszej witrynie artykule
"Czy strachoccy Piotrowscy są potomkami kniazia Iwaszki Kadyszewicza, wnuka tatarskiego księcia (mirzy) Najman-bega ... ?").
Oczekiwanie takie nie jest proste do spełnienia - nie mamy bezpośredniej informacji o genotypie kniazia Iwaszki ani innych historycznych członków rodziny tatarskich Piotrowskich. Nadzieje na pozytywny wynik naszego przedsięwzięcia opieraliśmy (opieramy) więc o założenie iż współcześnie żyją (zapewne żyją, nie wiemy tego na pewno) inni potomkowie Najman Bega, z rodzin o poświadczonym takim pochodzeniu. Jeżeli Piotrowscy ze Strachociny także pochodzą od Najman Bega to haplotypy członków tych rodzin muszą być podobne do uzyskanego przez nas, być może wręcz z nim identyczne.
Zakładaliśmy zatem, że to co robimy to zaledwie pierwszy krok w stronę bardziej gruntowych badań polegających na dotarciu do haplotypu kogoś takiego i porównaniu go z uzyskanym przez nas haplotypem (będziemy go tu nazywać haplotypem Piotrowskich ze Strachociny).
Teraz już wiemy, że sprawa jest nieco bardziej skomplikowana.
Biorąc pod uwagę to że wiedza na temat badań genetycznych, w tym znajomość stosowanej w nich nomenklatury, nie jest powszechna (sami musieliśmy się przez ich zawiłości ze sporym trudem przebijać) informację o uzyskanych wynikach badań rozpoczynamy od podzielenia się tą szczupłą cząstką wiedzy jaką udało się nam jednak pozyskać.
Zacznijmy od nomenklatury:
|
haplotyp (z grec. haplous - prosty, pojedynczy) - tu (ma i inne znaczenia): indywidualny zestaw specyficznych wartości mutacji w określonych miejscach DNA |
Specyfikacja haplotypu polega na podaniu dla pewnej ilości miejsc (lokacji) na chromosomie liczby powtórzeń występujących w tych lokacjach STR-ów (krótkich fragmentów DNA). Im lokacji jest więcej tym opis haplotypu jest oczywiście dokładniejszy. Lokacje dla których wyznacza się te liczby (markery) nie są przypadkowe. Wybiera się takie lokacje w których występuje największa różnorodność międzyosobnicza wynikająca z większej niż dla innych lokacji skłonności do występowania mutacji. Daje to jak największe zindywidualizowanie opisu haplotypu, co jest tu kluczowe.
W badaniach genetycznych markery wyznacza się dla znormalizowanych lokacji (wyżej podano jak się zapisuje ich numeryczne "adresy") i dla konkretnych ich ilości. Najczęściej uwzględnia się zaledwie 9 lokacji bo to na ogół wystarcza (choć oczywiście nie zawsze, o czym się niebawem przekonamy).
Haplotyp Piotrowskich ze Strachociny podano nam w wersji z 23 lokacjami (i oczywiście tyluż markerami). Jest to bardzo dobra dokładność, niestety haplotypów wyznaczonych z taką dokładnością jest stosunkowo mało i stąd nie ma z czym ich porównywać.
Tabela 1. Wyniki analiz genetycznych loci STR chromosomu Y Piotrowskich ze Strachociny
| locus | "mężczyzna" |
| DYS576 | 19 |
| DYS389 I | 13 |
| DYS448 | 20 |
| DYS389 II | 30 |
| DYS19 | 16 |
| DYS391 | 11 |
| DYS481 | 23 |
| DYS549 | 12 |
| DYS533 | 12 |
| DYS438 | 11 |
| DYS437 | 14 |
| DYS570 | 21 |
| DYS635 | 24 |
| DYS390 | 25 |
| DYS439 | 10 |
| DYS392 | 11 |
| DYS643 | 10 |
| DYS393 | 13 |
| DYS458 | 15 |
| DYS385 | 11, 15 |
| DY456 | 15 |
| Y GATA H4 | 12 |
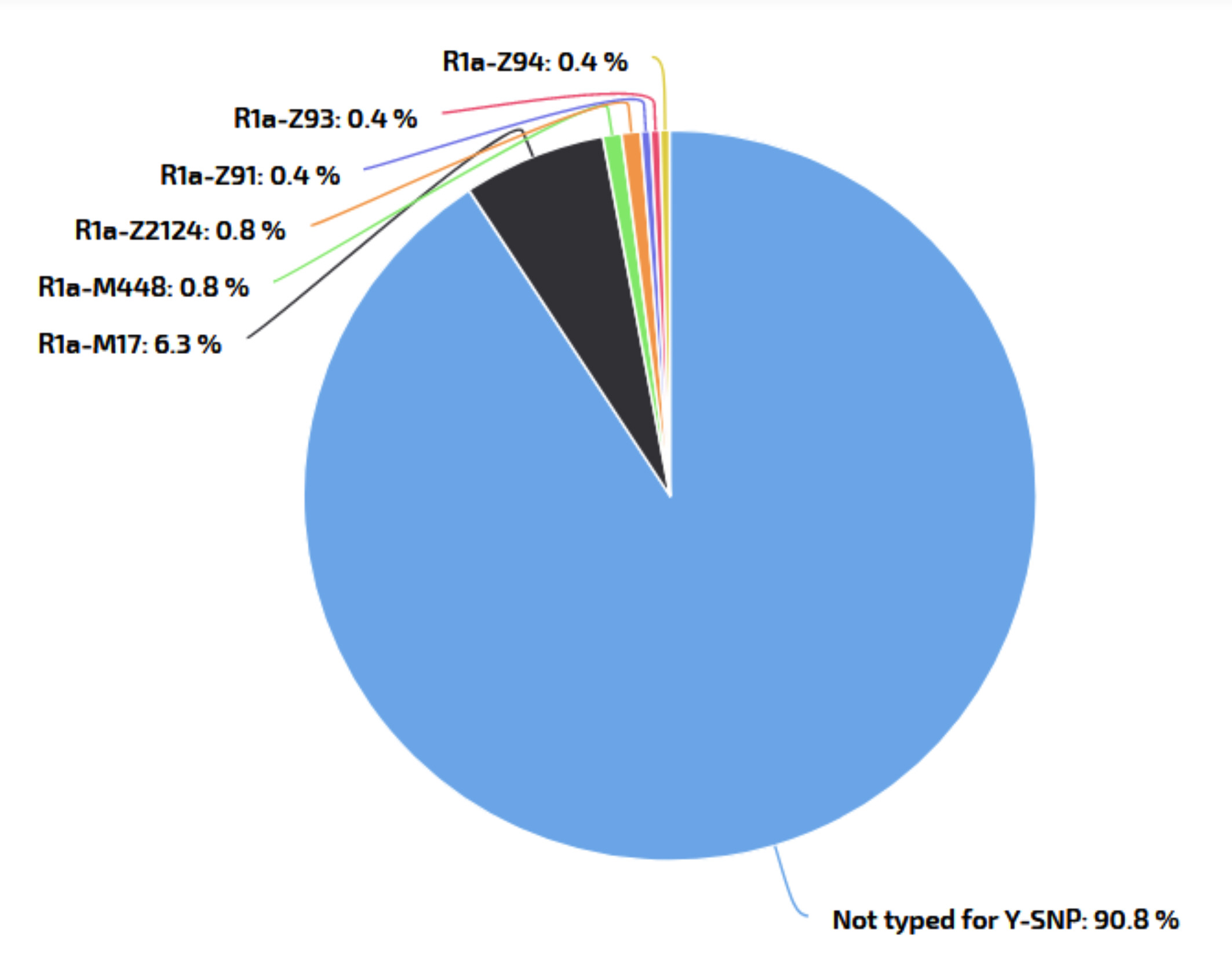
Sporządzony przez Instytut Genetyki Sądowej w Bydgoszczy raport z wyników zamówionej przez nas analizy zawiera przytoczone w Tabeli 1. dane podsumowane informacją: "Wyniki badań laboratoryjnych polegające na badaniu markerów STR na chromosomie Y, pozwalają na określenie przynależności badanej próbki do haplogrupy R1a".
Niestety, choć do haplogrupy R1a (uznawanej za "słowiańską" a nawet wręcz "polską") należy blisko 60% haplotypów Polaków jednak należy tu także ponad 30% haplotypów Tatarów (Kazańskich) oraz 50% Tatarów Polsko-Litewskich. W obu tych grupach ta haplogrupa jest również najliczniejsza. Jak u etnicznych Polaków.
Zatem przynależność do haplogrupy R1a nie daje żadnej podpowiedzi do odpowiedzi na pytanie czy przodkami ("po mieczu") Piotrowskich ze Strachociny w ogóle byli czy też nie byli Tatarzy.
Mogli być, mogli nie być.
Oprócz tego Raport zawiera jeszcze tylko szczegółowe informacje o tym kto, o jakich (w tym wypadku wysokich) kwalifikacjach i według jakiej procedury przeprowadził badania.
Także obszerny (16 stron A4) tekst na temat haplogrupy R1a, będący tłumaczeniem anglojęzycznego artykułu dostępnego w witrynie Eupedia
(https://www.eupedia.com/europe/Haplogroup_R1a_Y-DNA.shtml).
Tyle. Tylko tyle.
Na szczęście mamy Internet.
A w Internecie jest wszystko, można zatem znaleźć także bazy haplotypów umożliwiające porównania danego haplotypu z innymi.
Jedną z takich baz (prawdę mówiąc innej nie znamy, po znalezieniu tej jednej dalej już nie szukaliśmy) jest udostępniana bezpłatnie Y-Chromosome STR Haplotype Reference Database - YHRD firmowana przez International Society of Forensic Genetics (ISFG) i wspierana przez Promega Corporation.
Baza powstała jako efekt światowej współpracy genetyków, głównie zajmujących się zastosowaniami w kryminalistyce.
Jest stale powiększana, w czerwcu 2019 zawierała ponad 285 tysięcy próbek haplotypów pochodzących ze 135 krajów z całego świata, dziś pewno jest ich już więcej.
W zakładce "Database Composition" podawana jest informacja ile haplotypów pochodzi z którego państwa, ile z której metapopulacji, ile z jakiej haplogrupy i jakie lokacje STR-ów są brane pod uwagę w różnych wariantach specyfikacji haplotypów.
Wśród metapopulacji jakie uwzględnia baza najbardziej nas interesuje oczywiście metapopulacja Euroazjatycka (ogólnie) z podziałami na Ałtajską, Europejską, Hinduską, Indo-Irańską, Kaukaską i Uralsko-Jukagirską (Są też metapopulacje Afrykańska, Afro-Azjatycka, Wschodnioazjatycka, Australijsko-Aborygencka itd., itp., oczywiście także z dalszymi podziałami.).
O ile kryterium zaliczenia haplotypów do innych klas abstrakcji (państwo, haplogrupa, liczba uwzględnionych lokacji STR-ów) jest oczywiste to inaczej jest z zaliczaniem ich do metapopulacji. Prawdopodobnie robiono to najzupełniej uznaniowo. Między innymi wszystkich Tatarów z próbki pochodzącej z Białegostoku zaliczono do metapopulacji Euroazjatyckiej - Ałtajskiej, co wobec poświadczanej od dawna i przez liczne badania bardzo dużej, wynikającej z historycznych zaszłości, niehomogoniczności tej grupy etnicznej musi budzić mocno uzasadnione, wręcz oczywiste wątpliwości.
Baza informuje także skąd (z jakich państw, miast, instytucji, z jakich programów badawczych, wręcz wprost od kogo, z podaniem adresów mejlowych) pochodzą występujące w niej próbki haplotypów. Co mamy zamiar wykorzystać.
W sporządzonym dla nas przez Instytut Genetyki Sądowej w Bydgoszczy haplotypie Piotrowskich ze Strachociny uwzględniono 23 markery 22 lokacji STR-ów (23 markery bo jedna z lokacji jest podwójna). Taki wariant specyfikacji haplotypu nosi nazwę PowerPlex Y23 (człon „power” niewątpliwie wskazuje na „moc”, zatem w tym wypadku na precyzyjność). Wyspecyfikowanych w ten sposób (zatem w jakości PowerPlex Y23) haplotypów jest w bazie YHRD zaledwie 62 737. Nie ma wśród nich próbek pochodzących z Białorusi (także z Ukrainy), co, wobec dalszych wyników naszych analiz (zaraz się tu pojawią), wydaje się mieć bardzo istotne znaczenie.
Po wprowadzeniu danych i przeszukaniu przez program bazy nie znaleziono w niej ani jednego haplotypu identycznego z haplotypem Piotrowskich ze Strachociny. Czego się można było spodziewać (przypominamy, haplotypów w tak "mocnej" wersji z 23 markerami jest obecnie w bazie YHRD zaledwie ok. 63 tys.
Znalazł się natomiast jeden (tylko jeden) haplotyp nieomal identyczny ("1-step neighbor"). Ma on inną wartość markera STR tylko dla jednej lokacji. Konkretnie dla lokacji DYS458. Marker haplotypu Piotrowskich ze Strachociny ma tu wartość 15, natomiast ów "nieomal identyczny" haplotyp wartość 16. Oznacza to, że pewna maleńka sekwencja bazowych par nukleotydów w tym miejscu w jednym haplotypie powtarza się 15 a w drugim 16 razy. Reszta, zatem 22 markery STR, każdy z których może przyjmować dziesiątki różnych wartości (typowo 5 do 50), jest identyczna.
Nieistnienie mniej lub bardziej bliskiego pokrewieństwa osób o tych haplotypach wydaje się tu być wręcz niemożliwe, a przynajmniej skrajnie mało prawdopodobne (niewątpliwie wielokrotnie mniejsze niż szansa wygrania miliona w Totolotka gdzie chodzi o zgodność nie aż 22 lecz "zaledwie" 6 liczb). Gdyby osoba z tym "nieomal identycznym" haplotypem była dzieckiem dla którego matka szuka "płatnika alimentów" i gdyby sąd wystąpił o genetyczną ekspertyzę ojcostwa to prawdopodobieństwo że ojcem jest posiadacz haplotypu nazywanego tu haplotypem Piotrowskich ze Strachociny taka ekspertyza musiałaby oszacować na 99,55% (!) - taki wynik podaje analizujący bazę YHRD, dołączony do niej, program.
Posiadająca ten "nieomal identyczny" haplotyp osoba mieszka w Estonii i według danych z bazy YHRD należy do metapopulacji Uralsko-Jukagirskiej. Jukagirzy to maleńki naród z północno-wschodniej Syberii, jednak znacznie bardziej znanymi ludami należącymi do tej metapopulacji są pochodzący z okolic Uralu (stąd pierwsza część jej nazwy) Ugrofinowie, a wśród nich Węgrzy, Finowie, Estończycy ...
Próbki (w dokładności PowerPlex Y23) uznane w bazie za przynależne do metapopulacji Uralsko-Jukagirskiej (jest takich 1288) pochodzą przede wszystkim z Finlandii
(jest ich 1118, co stanowi ponad 86,8% wszystkich) oraz z Estonii (125, 9,7%), zaś reszta (45, 3,5%) z leżącej nad środkową Wołgą (tuż obok Tatarstanu) autonomicznej republiki Federacji Rosyjskiej Mari El. Jest raczej oczywiste, że "nasz" Estończyk nie jest Jukagirem lecz potomkiem Ugrofinów, którzy, jak wiemy do Europy przywędrowali przed wiekami z okolic Uralu. Wiemy jednak także, że część z tych Ugrofinów którzy pozostali na miejscu najpierw została Bułgarami (Wołżańskimi) a potem Tatarami (Kazańskimi) - niekiedy mówi się (rzecz jasna z grubą przesadą) iż Tatarzy Kazańscy to poturczeni Ugrofinowie właśnie.
To tyle.
Jak się wydaje tylko tyle daje się, w świetle tego co oferuje baza YHRD, odpowiedzialnie powiedzieć na temat haplotypu nazwanego tu przez nas haplotypem Piotrowskich ze Strachociny. Jest w niej zbyt mało próbek o takiej "rozdzielczości" jak "nasz" haplotyp by można było robić jakieś dalsze analizy.
Jednak w bazie YHRD są także dane "gorszej jakości". I jest ich dużo więcej. Między innymi znajdują się w niej pochodzące z Białegostoku próbki określone jako tatarskie. Wszystkie są w specyfikacji Minimum Y (z zaledwie 9 markerami STR).
Chcąc wykorzystać także takie dane z mniejszymi ilościami markerów STR powtórzyliśmy analizy odrzucając część markerów określającychych haplotyp Piotrowskich ze Strachociny i przedstawiając go w wariantach YFiler (16 markerów), PowerPlex Y (12 markerów) i Minimum Y (9 markerów) a następnie szukając w odpowiedniej części bazy identycznych haplotypów.
Wyniki uzyskane dla YFiler i PowerPlex Y nie rzucają na kolana.
Nadal z powodu szczupłości bazy danych dla tych wariantów specyfikacji haplotypów.
Dla wersji YFiler (16 markerów) znaleziono 8 trafień, 5 w próbkach pochodzących z Federacji Rosyjskiej i po jednym w danych pochodzących z Litwy, z Finlandii i z Niemiec (w bazie YHRD także tu nie ma próbek z Białorusi).
Dla wersji PowerPlex Y (12 markerów) znaleziono 127 trafień. Najwięcej bo aż 30 znowu w danych pochodzących z Federacji Rosyjskiej, 14 z Chin, 12 z Polski, po 10 z Białorusi i Litwy, po 6 z Niemiec i Indii, 4 z USA, 3 z Łotwy, 2 z Estonii itd.
Można to oczywiście jakoś interpretować lecz znacznie bardziej wiarygodne (i przekonywające) wnioski daje się wyprowadzać z analizy haplotypów w wersji Minimum Y (9 markerów). Dla tej wersji baza ma najwięcej próbek. Stąd skupimy się na tym wariancie.
W wariancie Minimum Y (który, przypominamy, uwzględnia zaledwie 9 markerów STR) w bazie YHRD występuje najwięcej bo aż 285406 próbek haplotypów z całego świata. Wśród nich jest aż 238 haplotypów identycznych z (okrojonym do wersji Minimum Y) haplotypem Piotrowskich ze Strachociny (aż 0,083%!, co domaga się i uwagi i jakiejś interpretacji).
Dla części (9,2%) z tych haplotypów program określił haplogrupę do której należą (jeżeli dobrze rozumiemy to dla pozostałych próbek nie było potrzebnych dodatkowych danych, dodatkowych bo przecież w zakresie specyfikacji haplotypu Minimum Y wszystkie te próbki są identyczne):

Jest zgoła oczywiste że szansa na znalezienie identycznego haplotypu i przypisanie go do jakiejś populacji zależy od liczby próbek znajdujących się w bazie. Próbek pochodzących z różnych populacji i z różnych państw. W bazie YHRD ilości te są niekiedy nader szczupłe (z takiej Kanady nie ma ani jednej!) i bardzo różne dla różnych państw. Z Chin pochodzi ok. 90 tys. próbek haplotypów (aż 22% wszystkich próbek!), z USA ok. 42 tys., z Polski ok. 8 tys., z Federacji Rosyjskiej ok. 4 tys., zaś z Białorusi zaledwie 498. Trudno tu znaleźć jakąś "mocną" prawidłowość ale tak czy inaczej chcąc wyprowadzać z powyższych danych jako tako wiarygodne wnioski musi się ten czynnik koniecznie uwzględniać.
Zrobiliśmy to dla wariantu Minimum Y którym się teraz zajmujemy. Zamiast brać pod uwagę bezwzględną liczbę występowań danego haplotypu w próbkach pochodzących z danego kraju obliczyliśmy względną częstotliwość jego występowania odniesioną do istniejących w bazie YHRD próbek haplotypów z niego pochodzących.
Rozkład trafień, zatem rozkład występowania haplotypów identycznych z haplotypem Piotrowskich ze Strachociny (w wersji Minimum Y) pomiędzy kraje przedstawia tabela 2. (uwzględniono w niej tylko te państwa dla których wystąpiło więcej niż 5 trafień). W trzecim wierszu tabeli podano (procentowo) obliczoną względną częstotliwość występowania tego haplotypu wśród istniejących w bazie YHRD haplotypów Minimum Y z danego kraju.
Tabela 2. Liczby haplotypów identycznych z haplotypem Piotrowskich ze Strachociny
(w wersji Minimum Y)
| Rosja | Polska | Białoruś | Chiny | Niemcy | Litwa | Ukraina | USA | India | Iran |
| 53 | 42 | 15 | 15 | 13 | 12 | 10 | 10 | 11 | 6 |
| 1,34% | 0,53% | 3,06% | 0,02% | 0,14% | 1,89% | 1,25% | 0,02% | 0,18% | 0,23% |
Tabela pokazuje, że najwięcej trafień, zatem najwięcej haplotypów identycznych z haplotypem Piotrowskich ze Strachociny (w okrojonej wersji) znowu znaleziono wśród haplotypów pochodzących z Federacji Rosyjskiej. Dopiero na drugim miejscu jest Polska. Wśród próbek pochodzących z Białorusi trafień jest kilkakrotnie mniej lecz to właśnie Białoruś jest bezapelacyjnym, absolutnym liderem pod względem procentowego udziału takich haplotypów w całej puli haplotypów pochodzących z danego kraju. Zdecydowanie wyprzedza tu Federację Rosyjską, Litwę, Ukrainę i Polskę, państwa z Białorusią sąsiadujące, dla których taki udział jest również wysoki jednak wyraźnie niższy.
Poszukano także haplotypów różniących się od haplotypu Piotrowskich ze Strachociny (w wersji Minimum) jedynie wartością jednego markera (1-step neighbors). Znaleziono ich aż 1335. Rozkład występowania takich haplotypów przedstawia tabela 3. (uwzględniono w niej tylko te państwa dla których ich procentowy udział jest większy niż 1%). Także tu takie haplotypy, zatem haplotypy genetycznie mniej lub bardziej bliskich krewniaków Piotrowskich ze Strachociny występują przede wszystkim na terenie Białorusi.
Tabela 3. Liczby haplotypów nieomal identycznych z haplotypem Piotrowskich ze Strachociny
(w wersji Minimum Y)
| Białoruś | Litwa | Rosja | Ukraina | Polska | Słowacja | India | India | Pakistan | Czechy |
| 29 | 36 | 10 | 192 | 26 | 212 | 29 | 110 | 35 | 32 |
| 5,93% | 5,68% | 5,08% | 3,24% | 3,24% | 2,41% | 2,41% | 1,84% | 1,53% | 1,37% |
Mocno potwierdza to hipotezę o pochodzeniu strachockich Piotrowskich z terenu dzisiejszej Białorusi, z pogranicza Litwy i Białorusi.
Ze stron w których zamieszkiwali historyczni tatarscy Piotrowscy, potomkowie kniazia Iwaszki, wnuka Najman Bega.
Wiemy o nich iż byli współwłaścicielami Sielec, małej wioski w powiecie oszmiańskim, obecnie znajdującej się właśnie na terenie Białorusi. Opuścili ją w pierwszej połowie XVII wieku gdy ostatni znany historykom przedstawiciel tego rodu (Alej) wyjechał "gdzieś w świat", nie wiadomo gdzie. Piotrowscy ze Strachociny utrzymują, że na Ukrainę, z której później albo on sam albo jego potomkowie uciekli w czasach Powstania Chmielnickiego. Uciekli pośpiesznie, zapewne "w ostatniej koszuli" chroniąc się przed niechybną śmiercią z rąk zrewoltowanej przez Chmielnickiego kozackiej czerni. Wraz z całą falą innych uchodźców, drobną polską szlachtą trafili na tereny dzisiejszego Podkarpacia, w przypadku Piotrowskich konkretnie do królewskiej wsi Strachocina, wówczas nieomal zupełnie wyludnionej.
Podsumowując: Nie mamy niepodważalnego genetycznego dowodu na pochodzenie strachockich Piotrowskich.
Jednak hipoteza że są potomkami kniazia Iwaszki, wnuka Najman Bega uzyskała nowe potwierdzenie.
Okazało się, że Piotrowscy ze Strachociny pochodzą stąd skąd muszą pochodzić by tymi potomkami mogli być.
Tak czy inaczej po pierwszym, już zrobionym kroku trzeba teraz zrobić krok kolejny: poszukać poświadczonych potomków Najman Bega należących do innych niż Piotrowscy linii rodowych i nakłonić któregoś z nich do zbadania swojego haplotypu.
Co z pewnością nie będzie łatwe.
Aha! Teraz już wiemy, że ten haplotyp musi być w wersji z możliwie dużą liczbą lokacji STR-ów.
Postawione wyżej tezy są ostrożne, bardzo ostrożne. Po tzw. "dojrzałym namyśle" można nawet dojść do wniosku, że wręcz nadmiernie ostrożne.
Przynajmniej w odniesieniu do jednej z informacji jakie pozyskaliśmy.
Chodzi o opartą o zbieżność wartości aż 22 z 23 markerów haplotypu Piotrowskich ze Strachociny informację o "naszym" Estończyku. Ta aż się prosi o tezę "nieostrożną", przy czym zastanawiamy się czy na pewno jest ona tezą "nieostrożną".
Zgadza się, analizy genetyczne są analizami statystycznymi zaś znane próbki haplotypów, w naszym wypadku próbki tworzące bazę YHRD, w żadnym razie nie są reprezentatywne dla populacji z których pochodzą. Nie znalazły się w bazie w wyniku jakichś systematycznych, dbających o reprezentatywność badań ale dość przypadkowo.
Bardzo często w ramach badań mniejszości narodowych, na ogół niewielkich, statystycznie mało istotnych. Łatwo się o tym przekonać wchodząc na zakładkę
"Database Composition" bazy. Stąd ostrożność w statystycznym interpretowaniu tych danych jest jak najbardziej uzasadniona.
Inaczej jest z "Estończykiem", "naszym Estończykiem". Tu nie mamy do czynienia ze statystyką, tu chodzi o jednostkowy fakt.
Statystyka pojawia się dopiero wtedy gdy stwierdzamy, że przy tak małej (zaledwie w jednym z 23 markerów!) różnicy między haplotypami szansa na czystą przypadkowość a nie bliskie pokrewieństwo (w lini męskiej) jest praktycznie równa zeru (szacując z grubsza szansa ta ma liczbowo wartość ok. 10-20, 1 podzielone przez liczbę składającą się z 1 i 20 zer, jest zatem ok. 10 miliardów, 1010 razy mniejsza niż szansa trafienia "szóstki" w totolotka). To owszem jest statystyka ale nie dotyczy ona analizowanych danych lecz tylko struktury tych danych (więc niejako samej, niezależnej od nich matematyki). Bardzo bliskie pokrewieństwo "naszego Estończyka" i Piotrowskich ze Strachociny można śmiało uznać za oczywisty, dobrze potwierdzony fakt.
Zaś o Estończyku, "naszym Estończyku" wiemy przecież to iż pochodzi z Metapopulacji Uralsko-Jukagirskiej, zatem jego przodkami są niewątpliwie Ugrofinowie, co skądinąd doskonale pasuje do tego co wiemy o pochodzeniu Estończyków. Czyż więc teza że jego bliscy krewniacy Piotrowscy ze Strachociny również pochodzą (w linii męskiej) od Ugrofinów jest naprawdę „nieostrożna”?
Tak czy inaczej ten fakt, fakt iż naszemu bliskiemu ("po mieczu") krewniakowi przypisuje się pochodzenie Uralsko-Jukagirskie jest i "sam w sobie" czymś istotnym i otwiera pole do różnych sensownych spekulacji.
Teraz trochę historii.
W VII w. na leżących na północ od Morza Czarnego Stepach Pontyjskich istniało duże, silne turkijskie państwo Wielka Bułgaria. Po roku 660 uległo ono podziałowi pomiędzy synów zmarłego wówczas władcy, potem korzystając z tego podbili je Chazarowie, zaś owi synowie wywędrowali ze swoimi ludźmi w różne strony świata. Trzech na zachód przy czym tylko jednemu z nich udało się odnieść znaczny sukces, podbił miejscową (słowiańską) ludność i stworzył państwo - Bułgarię (Dunajską). Po paru wiekach po rządzących nią turkijskich Bułgarach (musiało ich być niezbyt dużo) pozostała tylko nazwa i trochę osobliwości językowych.
Inny z braci poszedł na północ, w górę Wołgi. U jej zbiegu z rzeką Kamą założył państwo: Bułgarię Wołżańską. Tym razem Bułgarzy narzucili miejscowym nie tylko nazwę ale także język i sposób życia. Tymi "miejscowymi" byli zamieszkujący tamte tereny Ugrofinowie.
Bułgaria Wołżańska, ze swoją wysoką cywilizacją duchową i materialną istniała (i kwitnęła) przez kilka wieków. Także po tym gdy podbili ją Mongołowie i włączyli do swojego imperium. Do tej jego części która z czasem usamodzielniła się jako suwerenne państwo nazwane później Złotą Ordą. Tatarzy to mieszkańcy tego państwa.
Jest dość oczywiste, że część z nich, w szczególności ci którzy mieszkali na terenach dawnej Bułgarii Wołżańskiej byli genetycznymi potomkami dawnych Bułgarów Wołżańskich (dotyczy to zwłaszcza ich warstwy oświeconej), zatem w sporej części potomkami zbułgaryzowanych (poturczonych) Ugrofinów. Potwierdzają to współczesne badania genetyczne.
Niewątpliwie takie mogło być także pochodzenie genetyczne arystokraty Złotej Ordy Najman Bega. Sam chan Tochtamysz u którego pełnił on wysoką funkcję bega był potomkiem Czyngiz Chana ale działo się to blisko dwieście lat po mongolskim podboju gdy mongolskie geny nie były już warunkiem sine qua non bycia członkiem elity. Takie geny (a konkretnie pochodzący od Czyngiz Chana gen Y) były potrzebne by zostać chanem ale nie były niezbędne by sprawować wysokie funkcje a nawet rządzić (niekiedy mając za marionetkę formalnego chana). Przykładów jest wiele, z emirami Tamerlanem i (złotoordyńskim) Edigejem na czele.
Zatem potwierdzająca ich tatarskie pochodzenie informacja, że Piotrowscy ze Strachociny genetycznie wywodzą się z Tatarów Wołżańskich, nijak nie stoi w sprzeczności z przekonaniem iż są oni potomkami Najman Bega. Znaczyło by to tylko tyle, że i on był Tatarem pochodzącym właśnie z tej populacji.